ドラフト配列アセンブラーの研究開発で米国の後塵を拝している状況で考えるべきことの一つは、その先のテーマでしょう。約90%以上ヒトゲノム配列が確定した段階で考えられる面白いテーマとして遺伝子写像ソフトの作成があります。
この問題は昨年の演習3で課題として出しました。現在修士1年の小笠原準君が1年間かけて解決し、従来のソフトの100倍以上の効率向上を達成しました。その意味で既に解かれた問題ですが、ドラフト配列アセンブラー以上に今後重要になる技術なので再度説明し、課題として出します。
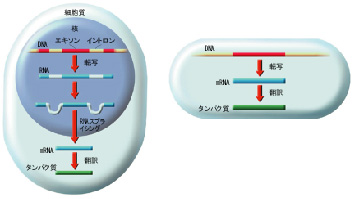
上図にしたがって、蛋白質が精製されるまでのプロセスを簡単に説明します。上図の左は、核をもつ真核生物(ヒトなど)における蛋白質精製の過程を示しています(右図は核のない原核生物の図で今回はあまり関係がないです)。核内にはDNAが存在し、その長さは塩基数にして、約2億6千万個(1番染色体)から約4500万個(22番染色体)と言われています。DNAからはその一部がまずRNAに転写されます。
問題1 どの部分が転写されるか否かが、解明されていない。
このRNAは核の外に出る際にRNAスプライシングという操作を受けて、イントロンと呼ばれる蛋白質精製には無駄な部分が除去されて、必要な部分であるエキソンと呼ばれる部分だけが append された結果が核外に出ます。、この append された配列を mRNA を呼びます。mRNAの長さは高々約1万個です。
問題2 RNA において、どこまでがエキソンで、どこからがイントロンか、解明されていない。mRNA を見ただけでは、エキソンの切れ目が分からない。
mRNA はその一部が蛋白質として翻訳されてゆきます。さて、現在までに蓄積された mRNA の総数は約330万個(3月時点)あり dbEST からすべて入手できます。またdbEST のデータは未整理であるため、少し整理が進んだデータが Unigene (約250万個登録 3月時点)から入手できます。しかし、これらのデータベースでは問題1&2を解決する情報が出ていません。
この問題を解決するには、各 mRNA の配列をDNA上にエキソンに分解しながら逆写像することが必要になります。この目的のために, est2genome (英国 Sanger Center, 1996年)、sim4 (Univ. of Penn. 1998 年) が開発したソフトがありましたが、長大なヒトゲノムに対しては使い物にならないスピードで動作します(我々が実験した限り1番染色体に対しては動作しなかった)。
そこで、長さ14の部分配列(長さは必ずしも14でなくてもよい)が、DNA配列上でどの位置に出現しているかを記述したインデックス表をあらかじめつくり、この表を引きながらエキソンの位置を推測し、かつエキソンが分離する境界では動的計画法により写像を精緻化するプログラムを小笠原君が開発しました。アルゴリズムの詳細は小笠原君の卒業論文(図書室にあります)を見てください。
写像した結果を参照することで問題1&2に関してかなり詳細な情報が大量に得られるようになりました。結果は以下のサイトから2001年1月より公開しています。
Gene Resource Locator http://grl.gi.k.u-tokyo.ac.jp/
写像結果を解析してヒト遺伝子の総数を予測するための機能や、RDB 化や Flash を使った動的な GUI を博士1年の本蔵君が作成しています。
実は小笠原君が写像ソフトを開発しているのと同時期に University of California, Santa Cruz の Jim Kent もかなり類似した方法でソフトを開発し、写像結果を以下のサイトから公開しています。
UCSC Human Genome Browser http://genome.cse.ucsc.edu/goldenPath/decTracks.html
全く同時期の開発なので、どちらが先か否かよくわからない状況ですが、全く異なるチームが、異なる実装を行い、殆ど同じ写像が得られているので、ゲノム解析にとっては写像の信頼性を増す効果がありました。
さて課題ですが、小笠原君の卒業論文を読んで、改良が加えられないか考えて、実装することです。
データソースをまとめると以下のようになります。
| ヒトゲノム配列(DNA配列) 801MB, 国際ヒトゲノムチーム |
http://genome.cse.ucsc.edu/goldenPath/12dec2000/bigZips/chromFa.zip |
| ヒトEST配列 467MB, 250万個, 重複有 | ftp://ncbi.nlm.nih.gov/repository/UniGene/Hs.seq.all.Z |
| ヒトEST配列 29.4MB, 重複なし,デバッグ用に適する | ftp://ncbi.nlm.nih.gov/repository/UniGene/Hs.seq.uniq.Z |
| マウスEST, 254MB, 重複有 | ftp://ncbi.nlm.nih.gov/repository/UniGene/Mm.seq.all.Z |
| ヒトEST配列 19.6MB, 重複なし,デバッグ用に適する | ftp://ncbi.nlm.nih.gov/repository/UniGene/Mm.seq.uniq.Z |
笠原君の考えている方法に参考になりそうな手法に suffix array があります。
「大規模テキスト索引(suffix array)の構築法とその情報検索への応用」 定兼邦彦(東北大学)
PPTファイル